- I. Tổng quan về vấn đề lâm sàng
- 1. Ảnh hưởng của bệnh thoái hóa cột sống
- 2. Bài toán đặt ra trong cuộc thi
- 3. Nhiệm vụ
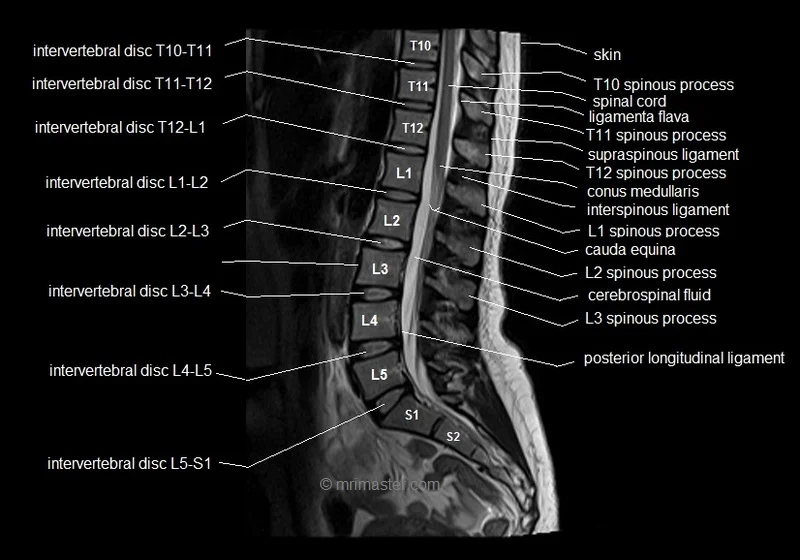
- 4. Tổng Quan Giải Phẫu Cột Sống
- 5. Tổng quan về Hẹp lỗ liên hợp (Foraminal Narrowing)
- 6. Tổng quan về Hẹp dưới khớp (Subarticular Stenosis)
- 7. Tổng quan về Hẹp ống sống (Canal Stenosis)
- 8. Tổng quan về hình ảnh y học (Imaging Overview)
- Xử lý dữ liệu
I. Tổng quan về vấn đề lâm sàng
1. Ảnh hưởng của bệnh thoái hóa cột sống
Các bệnh lý thoái hóa cột sống ảnh hưởng nghiêm trọng đến chất lượng sống của người bệnh. Việc phát hiện các tình trạng này là vô cùng quan trọng để xây dựng phác đồ điều trị phù hợp cho từng bệnh nhân. Do đó, phát triển các phương pháp để phát hiện và đánh giá mức độ nghiêm trọng của các tình trạng thoái hóa trên hình ảnh y học là rất cần thiết.
2. Bài toán đặt ra trong cuộc thi
Thử thách này tập trung vào việc phát hiện ba loại tổn thương chính ở vùng cột sống thắt lưng (lumbar) như sau (chi tiết về giải phẫu sẽ được đề cập bên dưới):
- Hẹp lỗ liên hợp (Foraminal narrowing): xảy ra ở bên trái hoặc phải tại một vị trí xác định trên cột sống.
- Hẹp dưới khớp (Subarticular stenosis): xảy ra ở bên trái hoặc phải tại một vị trí xác định.
- Hẹp ống sống (Canal stenosis): chỉ xảy ra tại một vị trí xác định. Mỗi tình trạng này có thể xuất hiện tại nhiều mức đốt sống khác nhau, cụ thể là tại từng đĩa đệm giữa các thân đốt sống (ví dụ: L4/5 là đĩa đệm giữa đốt sống L4 và L5).
3. Nhiệm vụ
Với mỗi tình trạng trên, chúng ta cần dự đoán mức độ chèn ép theo ba cấp độ:
- Bình thường/nhẹ (normal/mild)
- Trung bình (moderate)
- Nặng (severe) File ví dụ sample_submission.csv sẽ giúp hình dung rõ hơn về định dạng kết quả cần nộp. Đối với mỗi trường hợp (bệnh nhân, vị trí đốt sống, loại tổn thương, mức độ), chúng ta phải xuất ra một xác suất từ 0 đến 1, đại diện cho khả năng bệnh nhân đó thuộc từng mức độ tương ứng, tại từng vị trí và loại tổn thương. Cụ thể, bạn sẽ cần dự đoán xác suất cho từng mức độ (bình thường/nhẹ, trung bình, nặng) tại mỗi mức đốt sống:
- l1_l2
- l2_l3
- l3_l4
- l4_l5
- l5_s1
 cho từng tình trạng:
cho từng tình trạng: - spinal_canal_stenosis
- left_neural_foraminal_narrowing
- right_neural_foraminal_narrowing
- left_subarticular_stenosis
- right_subarticular_stenosis).
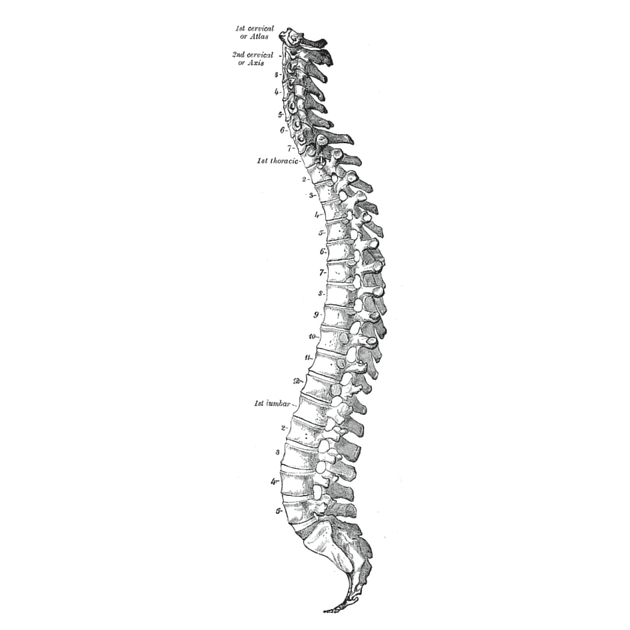
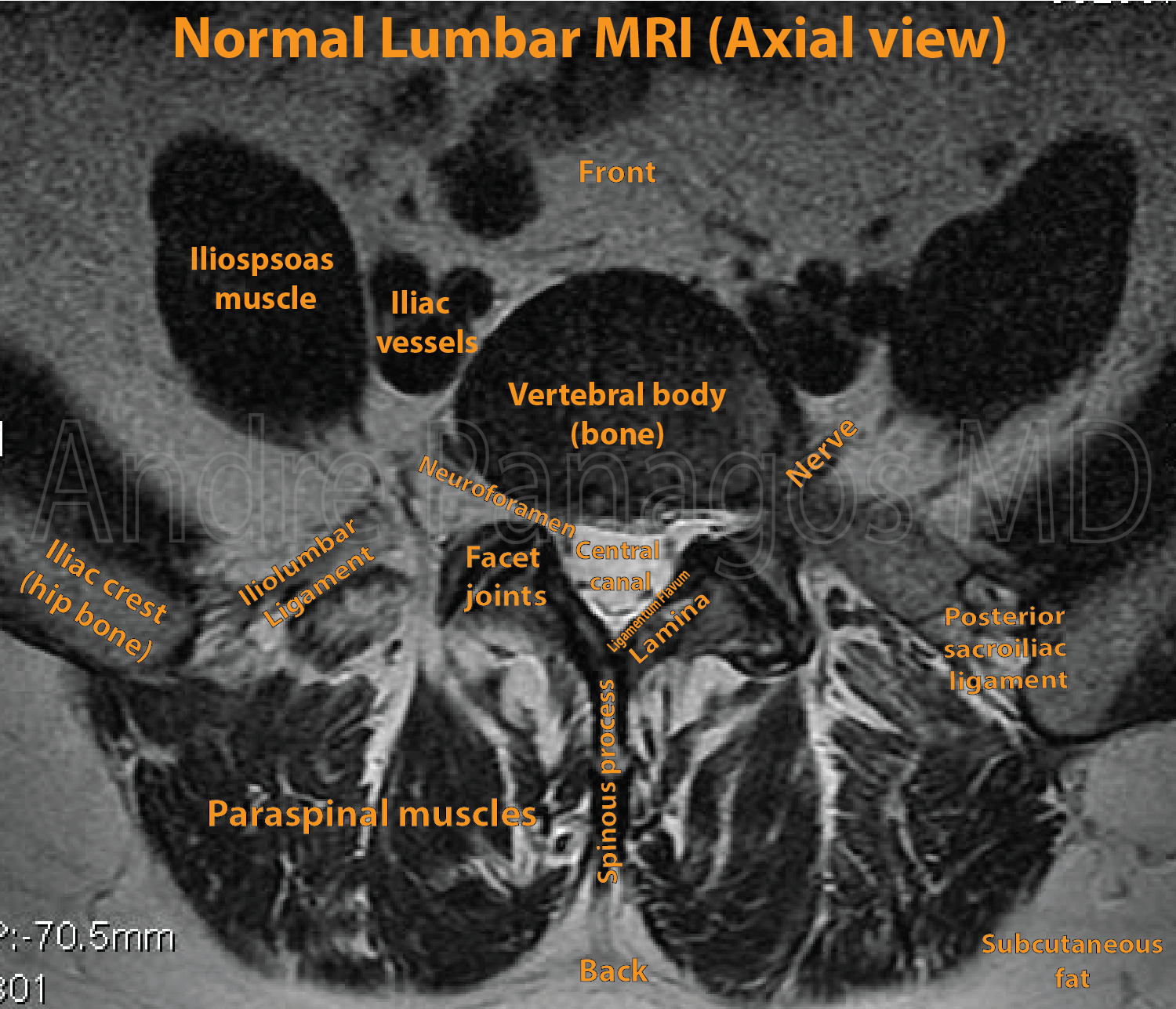
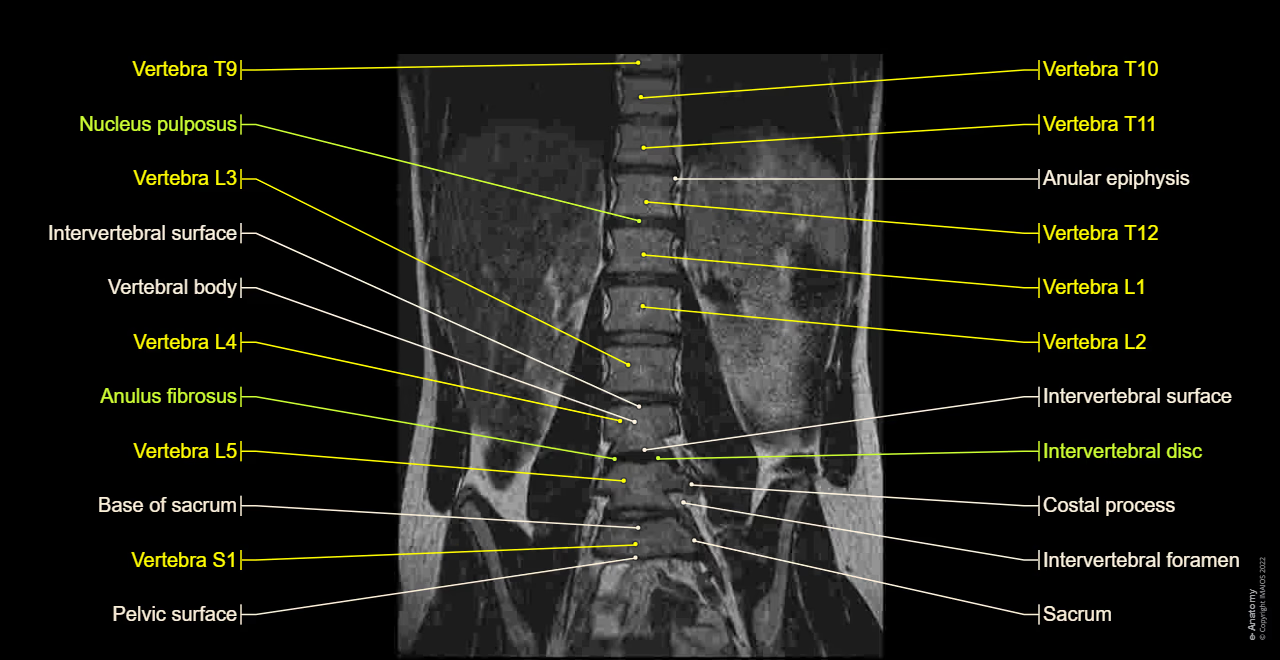
4. Tổng Quan Giải Phẫu Cột Sống
Cấu trúc các vùng của cột sống
Cột sống của con người được chia thành 4 vùng chính:
- Vùng cổ (cervical): gồm 7 đốt sống (C1–C7)
- Vùng ngực (thoracic): gồm 12 đốt sống (T1–T12)
- Vùng thắt lưng (lumbar): gồm 5 đốt sống (L1–L5)
- Vùng cùng (sacral): gồm 3 đến 5 đốt sống dính liền nhau thành một khối
Đĩa đệm và tủy sống
-
Đĩa đệm: Giữa mỗi thân đốt sống ở tất cả các vùng (trừ vùng cùng) đều có một đĩa đệm, giúp giảm xóc và linh hoạt cho cột sống.
-
Tủy sống: Chạy dọc phía sau các thân đốt sống, nằm trong ống sống, là nơi chứa các dây thần kinh quan trọng.
-
Tại mỗi thân đốt sống, các dây thần kinh cột sống sẽ rời khỏi tủy sống thông qua các lỗ gọi là lỗ liên hợp (foramina) nằm giữa hai đốt sống liền kề.
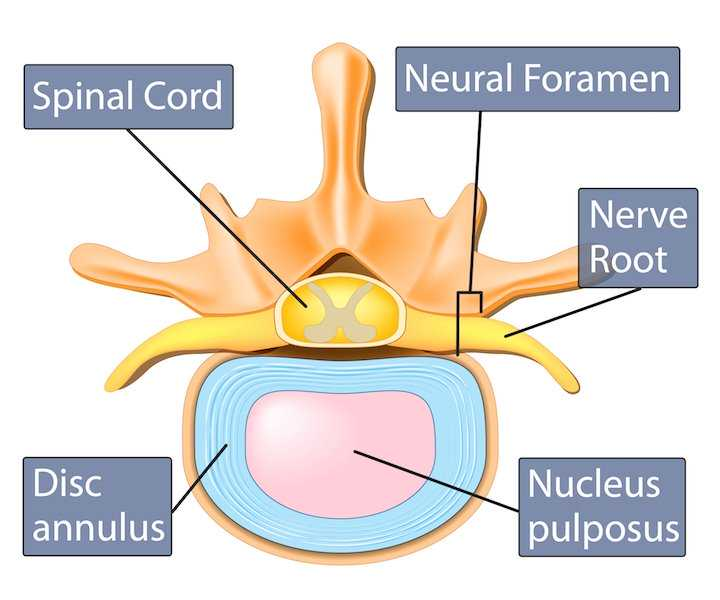
-
Spinal Cord: Tủy sống
-
Neural Foramen: Lỗ liên hợp (lỗ thần kinh)
-
Nerve Root: Rễ thần kinh
-
Disc annulus: Vòng sợi đĩa đệm
-
Nucleus pulposus: Nhân nhầy (nhân tủy) đĩa đệm
Nguyên nhân gây chèn ép dây thần kinh/tủy sống
- Chèn ép tủy sống hoặc dây thần kinh có thể gây ra các cơn đau dữ dội cho người bệnh.
- Những nguyên nhân thường gặp gồm:
- Phình đĩa đệm: Đĩa đệm bị lồi ra ngoài, chèn vào dây thần kinh/tủy sống.
- Thoái hóa xương: Các biến đổi ở xương do lão hóa làm xương phát triển gai xương hoặc bị lún, gây chèn ép.
- Chấn thương: Tác động lực mạnh làm biến dạng cấu trúc cột sống.
- Dày dây chằng: Dây chằng quanh tủy sống bị dày lên, thu hẹp không gian cho tủy/dây thần kinh.
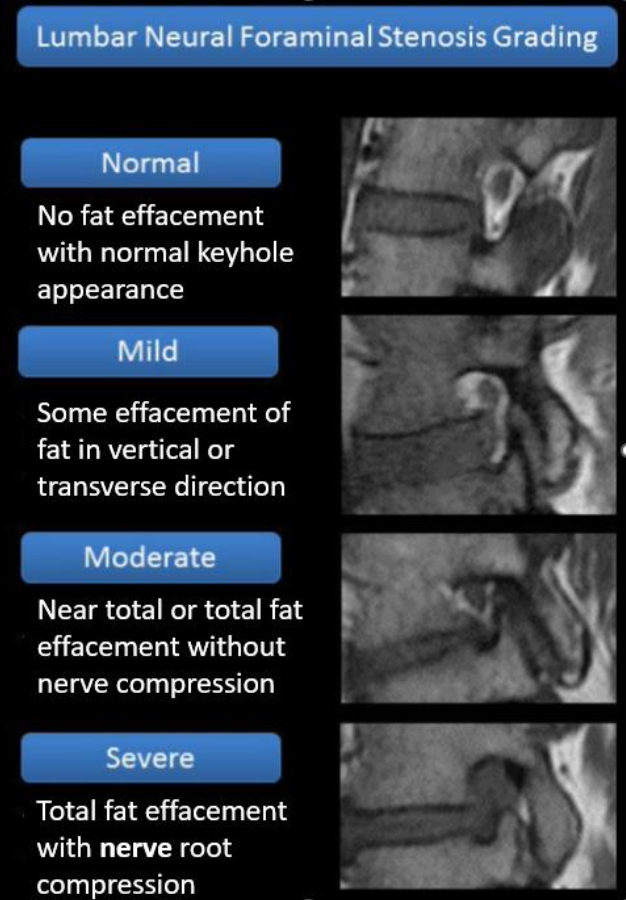
5. Tổng quan về Hẹp lỗ liên hợp (Foraminal Narrowing)
Định nghĩa & vị trí
- Tủy sống có các dây thần kinh đi ra ngoài qua các lỗ gọi là lỗ liên hợp (foramina).
- Các lỗ này quan sát rõ nhất trên hình ảnh MRI ở mặt phẳng sagittal (mặt cắt dọc bên).
- Trong một số trường hợp, các lỗ này bị chèn ép (hẹp lại), gây nên tình trạng hẹp lỗ liên hợp.
- Khi lỗ liên hợp bị hẹp, các rễ thần kinh bị chèn ép.
- Điều này gây ra đau theo vùng chi phối của dây thần kinh phía sau vị trí bị chèn ép (thường là đau lan xuống chân, hoặc yếu cơ).
Minh họa và tiêu chuẩn phân loại mức
-
Ảnh MRI mặt cắt sagittal sẽ cho thấy rõ các lỗ liên hợp.
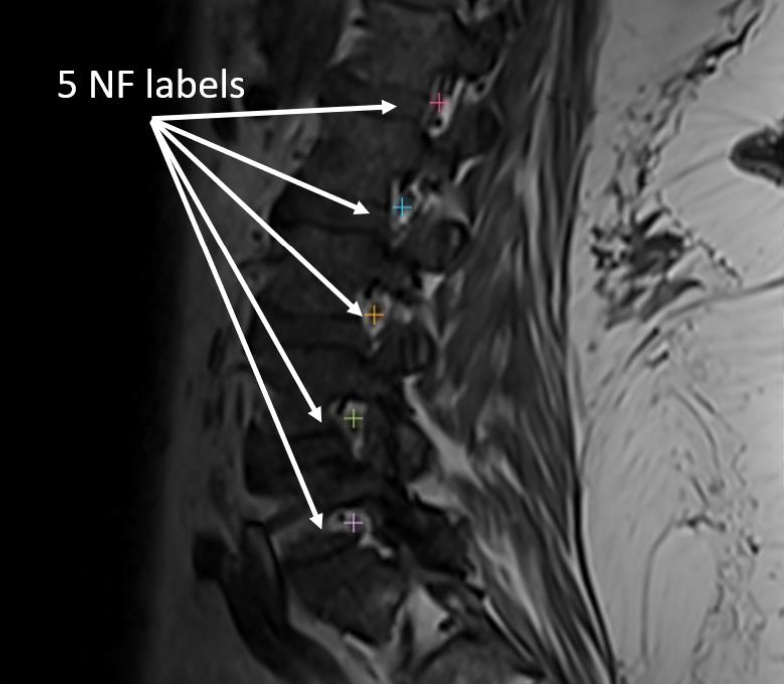
-
Ảnh bên trái minh họa lát cắt MRI với các lỗ liên hợp được đánh dấu bằng dấu cộng.
-
Ảnh bên phải minh họa tiêu chuẩn phân loại mức độ chèn ép (grading criteria):
-
Normal/Mild (Bình thường/nhẹ)
-
Moderate (Trung bình)
-
Severe (Nặng)
(Lưu ý: Trong thử thách này, Normal/Mild được gộp thành một nhãn duy nhất.)
-
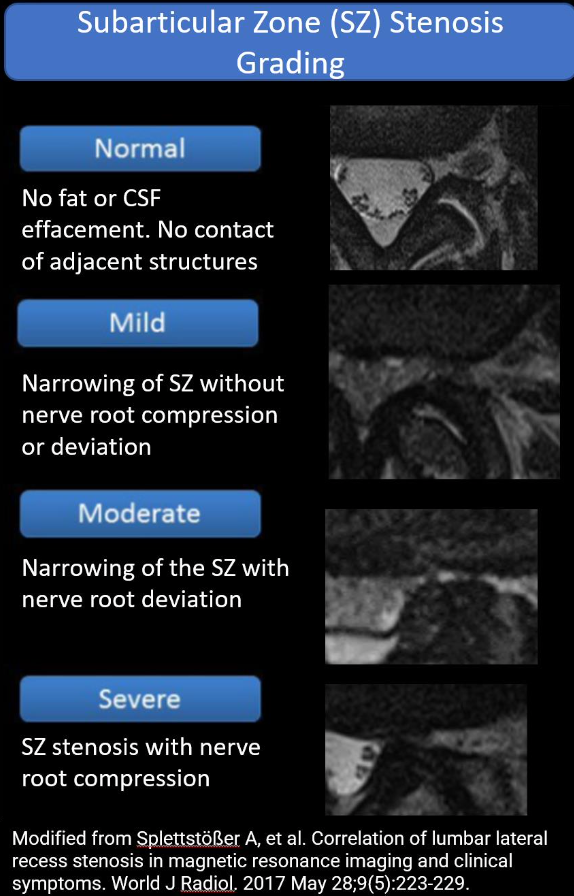
6. Tổng quan về Hẹp dưới khớp (Subarticular Stenosis)
Định nghĩa & vị trí
- Hẹp dưới khớp là tình trạng chèn ép tủy sống xảy ra tại vùng dưới khớp (subarticular zone) của ống sống.
- Sự chèn ép này quan sát rõ nhất trên ảnh MRI mặt phẳng axial (mặt cắt ngang).
Minh họa vùng giải phẫu liên quan
- Ảnh bên phải là sơ đồ minh họa vị trí vùng dưới khớp nơi thường xảy ra chèn ép.
- Vùng này nằm ngay sát bên dưới diện khớp của các đốt sống, nơi dây thần kinh dễ bị chèn ép do các thay đổi thoái hóa hoặc phình đĩa đệm.
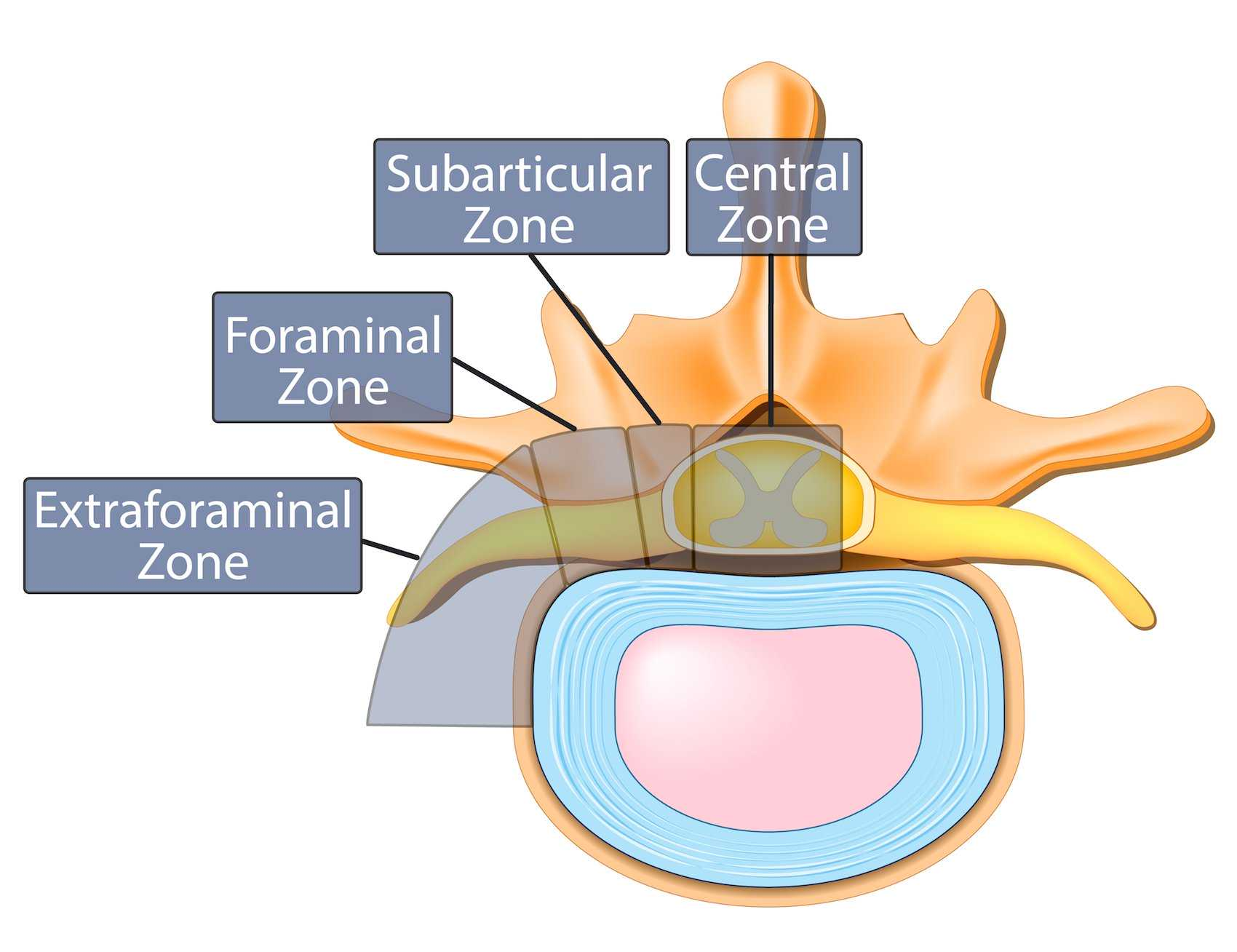
-
Subarticular Zone: Vùng dưới khớp
-
Central Zone: Vùng trung tâm
-
Foraminal Zone: Vùng lỗ liên hợp
-
Extraforaminal Zone: Vùng ngoài lỗ liên hợp
-
Ảnh bên phải minh họa tiêu chuẩn đánh giá mức độ hẹp dưới khớp:
-
Normal/Mild (Bình thường/nhẹ)
-
Moderate (Trung bình)
-
Severe (Nặng)
-
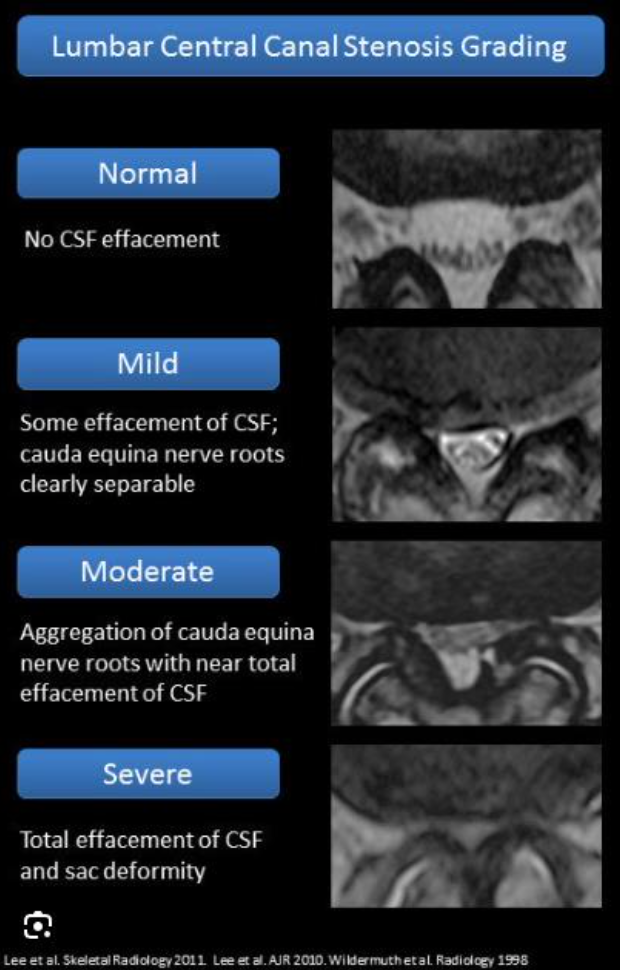
7. Tổng quan về Hẹp ống sống (Canal Stenosis)
Định nghĩa & nguyên nhân
- Hẹp ống sống là tình trạng bị chèn ép trong ống sống – nơi tủy sống đi qua.
- Các nguyên nhân phổ biến gây chèn ép gồm:
- Phình đĩa đệm: Đĩa đệm bị lồi, đẩy vào ống sống.
- Chấn thương: Tác động làm biến dạng, thu hẹp lòng ống sống.
- Gai xương (osteophyte): Gai xương phát triển do thoái hóa các đốt sống.
- Dày dây chằng: Dây chằng chạy dọc ống sống bị dày lên.
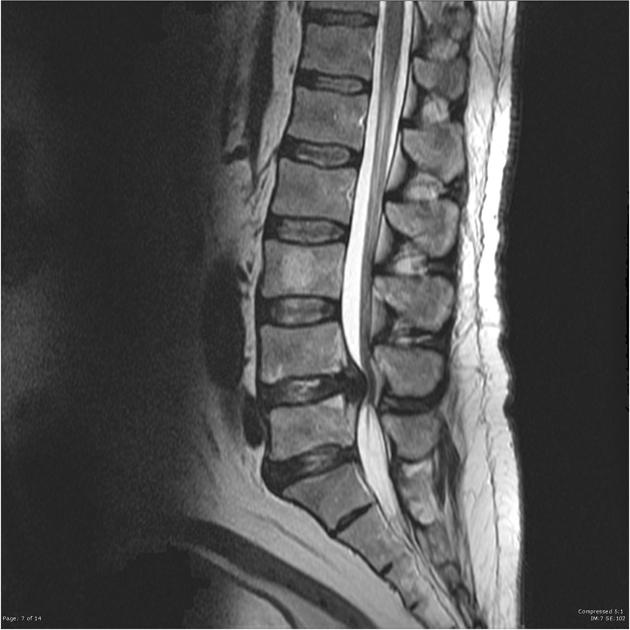
Hình ảnh & cách đánh giá mức độ hẹp
- Mức độ chèn ép trong ống sống thường được đánh giá rõ nhất trên ảnh MRI mặt phẳng axial (mặt cắt ngang).
- Ảnh bên phải minh họa vùng bị hẹp ống sống trên mặt phẳng sagittal (mặt cắt dọc bên) để giúp dễ hình dung tổng thể vị trí.
- Ảnh bên phải là tiêu chuẩn phân loại mức độ hẹp ống sống:
- Normal/Mild (Bình thường/nhẹ)
- Moderate (Trung bình)
- Severe (Nặng)
8. Tổng quan về hình ảnh y học (Imaging Overview)
Các mặt phẳng chụp MRI cột sống
-
Axial plane: Cắt cơ thể thành các lát ngang, giống như các lát bánh mì được cắt từ trên xuống dưới, vuông góc với trục cột sống.
-
Sagittal plane: Cắt cơ thể theo chiều dọc từ trái sang phải, song song với trục của cột sống.
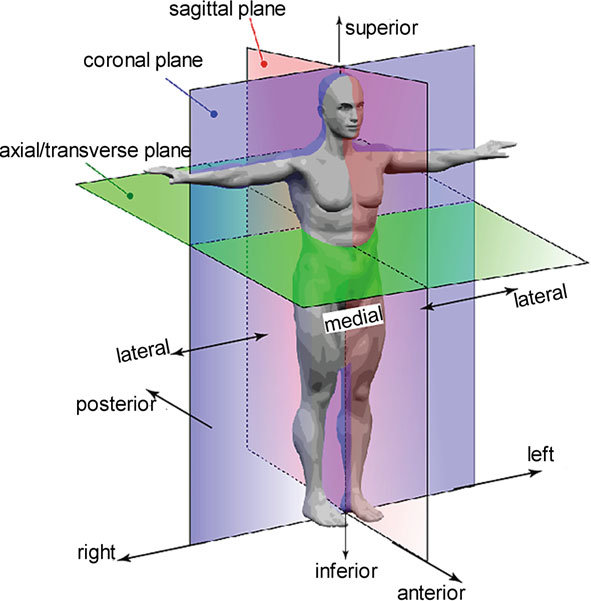
-
MRI cột sống có thể được chụp ở ba mặt phẳng chính:
- Mặt phẳng axial (ngang): Lấy lát cắt song song với mặt đất, vuông góc với trục cột sống, chia cơ thể thành phần trên và dưới.
- Mặt phẳng sagittal (dọc bên): Lấy lát cắt song song với trục cột sống, chia cơ thể thành bên trái và bên phải.
-
Mặt phẳng coronal (trán): Lấy lát cắt chia cơ thể thành mặt trước và mặt sau.
-
Trong thử thách này, chúng ta chủ yếu cần sử dụng hai loại mặt phẳng:
- Axial (ngang)
- Sagittal (dọc bên)
Các loại ảnh MRI
-
MRI có nhiều biến thể, nhưng cơ bản chia thành hai loại chính:
- T1 weighted (T1W):
- Mô mỡ hiển thị sáng hơn.
- Phần trong của xương (chứa mỡ tủy xương) sẽ sáng trên ảnh T1.
- T2 weighted (T2W):
- Nước (dịch) hiển thị sáng hơn.
- Ống sống và các vùng chứa dịch sẽ sáng trên ảnh T2.
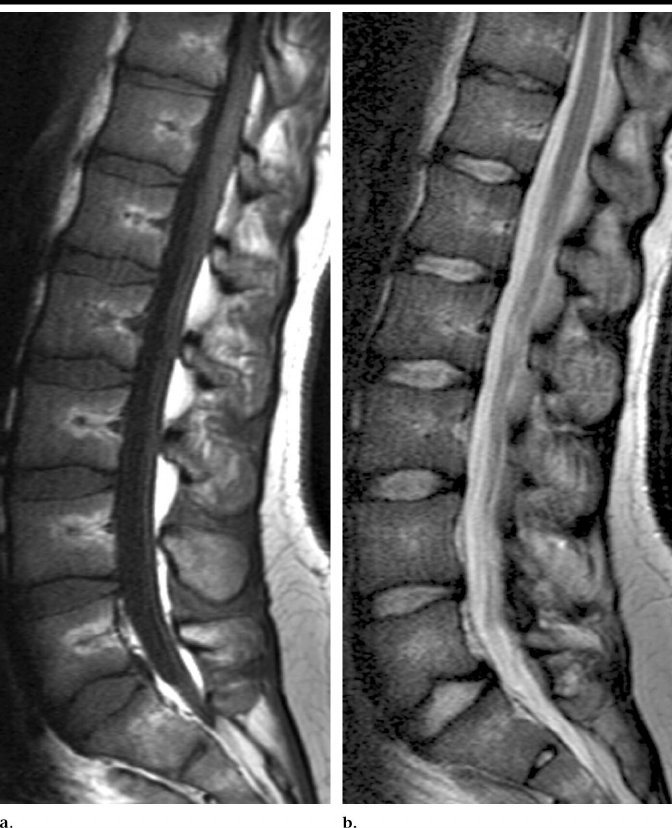
T1W trái và T2W phải
- T1 weighted (T1W):
4. Đặc điểm cần lưu ý khi xử lý ảnh MRI
- Ảnh MRI không được chuẩn hóa tuyệt đối về giá trị pixel (khác với ảnh CT). → Nghĩa là giá trị sáng/tối trên ảnh MRI có thể khác nhau tùy từng máy, từng lần chụp.
- Khi xây dựng mô hình, bạn có thể cần chuẩn hóa lại ảnh (hoặc không, tùy cách tiếp cận của bạn).
Xử lý dữ liệu
1. Cấu trúc thư mục dữ liệu
.
├── ExploreData.ipynb # Notebook phân tích dữ liệu (file bạn đang mở)
└── /kaggle/input/rsna-2024-lumbar-spine-degenerative-classification/
├── test_images/
│ ├── 1005139/
│ │ └── 609308237/
│ │ ├── 1.dcm
│ │ └── ...
│ └── ...
├── test_series_descriptions.csv
├── train_images/
│ ├── 4003253/
│ │ └── 702807833/
│ │ ├── 1.dcm
│ │ └── ...
│ └── ...
├── train_label_coordinates.csv
├── train_series_descriptions.csv
└── train.csv- train_images/ và test_images/: Thư mục chứa ảnh MRI theo từng bệnh nhân và từng series (mỗi file
.dcmlà một lát cắt MRI). - train.csv: File chứa nhãn mức độ thoái hóa cho từng trường hợp.
- train_label_coordinates.csv: Chứa tọa độ vùng tổn thương trên ảnh.
- train_series_descriptions.csv / test_series_descriptions.csv: Mô tả thông tin chi tiết về từng series ảnh MRI.
2. Tải thông tin chẩn đoán (Loading Diagnosis Information)
Tải dữ liệu thông tin chẩn đoán trong bộ dữ liệu và xem tổng quan về phân bố các trường hợp bệnh.
import pandas as pd
import matplotlib.pyplot as plt
import cv2
import pydicom
train = pd.read_csv('/kaggle/input/rsna-2024-lumbar-spine-degenerative-classification/train.csv')
print("Total Cases: ", len(train))
# Output: Total Cases: 1975
train.columnsPhân tích phân bố các loại chẩn đoán
import numpy as np
import os
import glob
from tqdm import tqdm
import warnings
figure, axis = plt.subplots(1, 3, figsize=(20, 5))
for idx, d in enumerate(['foraminal', 'subarticular', 'canal']):
diagnosis = list(filter(lambda x: x.find(d) > -1, train.columns))
dff = train[diagnosis]
with warnings.catch_warnings():
warnings.simplefilter(action='ignore', category=FutureWarning)
value_counts = dff.apply(pd.value_counts).fillna(0).T
value_counts.plot(kind='bar', stacked=True, ax=axis[idx])
axis[idx].set_title(f'{d} distribution')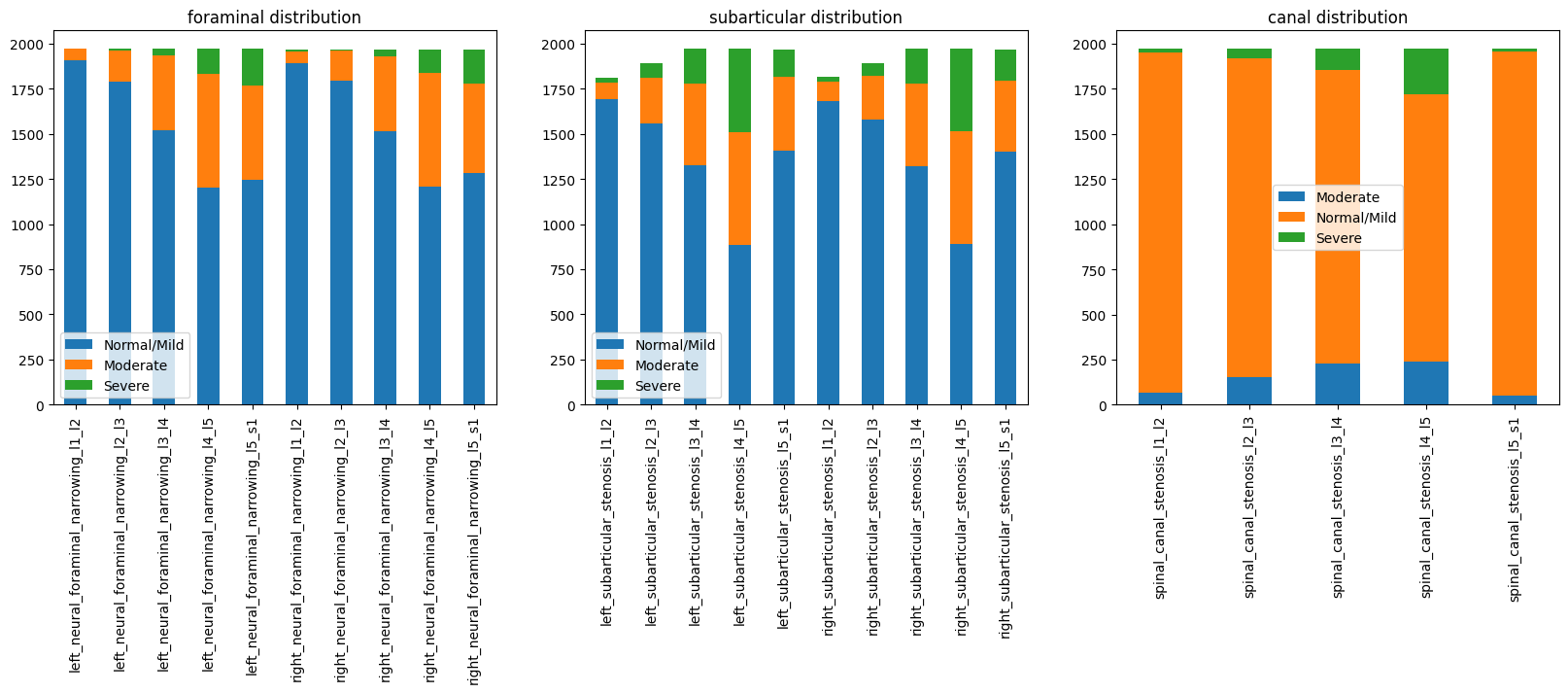
- Foraminal narrowing (Hẹp lỗ liên hợp)
- Subarticular stenosis (Hẹp dưới khớp)
- Canal stenosis (Hẹp ống sống) Nhận xét:
- Phần lớn bệnh nhân có mức độ bình thường/nhẹ (normal/mild) ở tất cả các loại tổn thương.
- Ở một số trường hợp (đặc biệt là subarticular stenosis), dữ liệu bị thiếu (missing data).
- Nguyên nhân: Một số ảnh không ghi lại được đầy đủ vùng cột sống (đặc biệt là các đốt sống phía trên cùng – “most superior vertebral bodies” – ít khi nằm trọn trong trường quan sát của máy MRI).
3. Cấu trúc folder ảnh MRI (Loading in images)
Sau khi đã biết tổng quan về phân bố các chẩn đoán, bây giờ ta sẽ tải và xem ví dụ một bộ ảnh MRI của một bệnh nhân.
- Mỗi bệnh nhân sẽ có một Study (được định danh bởi
StudyInstanceUID). - Mỗi Study lại có thể chứa nhiều Series (được định danh bởi
SeriesInstanceUID). - Mỗi Series là một bộ ảnh (scan) theo một mặt phẳng hoặc tham số kỹ thuật nhất định, và bên trong Series là nhiều ảnh (mỗi ảnh đại diện cho một lát cắt), mỗi ảnh có
SOPInstanceUIDriêng. Tóm tắt cấu trúc: - Bệnh nhân → Study (StudyInstanceUID)
- Series (SeriesInstanceUID)
- Ảnh/lát cắt (SOPInstanceUID) Tất cả thông tin này được liên kết với nhau thông qua các file CSV:
- Series (SeriesInstanceUID)
- File CSV chứa nhãn (chẩn đoán) đã nói ở phần trước.
- File metadata DICOM riêng biệt chứa thông tin mô tả từng series, ví dụ như:
- Loại mặt phẳng: sagittal (dọc), axial (ngang),…
- Loại chuỗi: T1 hay T2 weighted
- Lưu ý: Tên của các chuỗi mô tả series (series descriptions) không hoàn toàn chuẩn hóa, nên có thể cần xử lý thêm khi phân tích.
Trích xuất metadata cho từng scan MRI
Với mỗi scan (mỗi bệnh nhân), ta sẽ tạo một đối tượng (meta_obj) chứa thông tin cơ bản sau:
meta_obj = {
StudyInstanceUID: {
'folder_path': ... # Đường dẫn tới thư mục ảnh của study đó
'SeriesInstanceUIDs': [...] # Danh sách các SeriesInstanceUIDs (các series con)
'SeriesDescriptions': [...] # Danh sách mô tả của từng series (ví dụ: Sagittal T2, Axial T1, ...)
}, ...
}a. Liệt kê tất cả các study (bệnh nhân)
- os.listdir để lấy danh sách thư mục ứng với từng StudyInstanceUID (bỏ qua file hệ thống .DS_Store nếu có trên Mac).
# List out all of the Studies we have on patients.
part_1 = os.listdir('/kaggle/input/rsna-2024-lumbar-spine-degenerative-classification/train_images')
part_1 = list(filter(lambda x: x.find('.DS') == -1, part_1))b. Đọc file metadata mô tả các series
- Đọc file CSV: train_series_descriptions.csv để lấy thông tin chi tiết về mô tả series cho từng study và series.
df_meta_f = pd.read_csv('/kaggle/input/rsna-2024-lumbar-spine-degenerative-classification/train_series_descriptions.csv')c. Tạo đối tượng meta_obj
- Với mỗi study:
- Lưu đường dẫn thư mục ảnh (
folder_path) - Lưu danh sách series con (
SeriesInstanceUIDs) bên trong folder study đó.
- Lưu đường dẫn thư mục ảnh (
p1 = [(x, f"/kaggle/input/rsna-2024-lumbar-spine-degenerative-classification/train_images/{x}") for xin part_1]
meta_obj = { p[0]: { 'folder_path': p[1],
'SeriesInstanceUIDs': []
}
for p in p1 }for m in meta_obj:
meta_obj[m]['SeriesInstanceUIDs'] = list(
filter(lambda x: x.find('.DS') == -1,
os.listdir(meta_obj[m]['folder_path'])
)
)d. Ghép mô tả series (series description) cho từng series
- Với mỗi series trong từng study, tìm mô tả phù hợp trong file CSV (dựa vào
study_idvàseries_id). - Thêm vào trường SeriesDescriptions (danh sách mô tả kiểu ảnh, ví dụ: Sagittal T2/STIR, Axial T2, Sagittal T1,…).
# grabs the correspoding series descriptions
for k in tqdm(meta_obj):
for s in meta_obj[k]['SeriesInstanceUIDs']:
if 'SeriesDescriptions' not in meta_obj[k]:
meta_obj[k]['SeriesDescriptions'] = []
try:
meta_obj[k]['SeriesDescriptions'].append(
df_meta_f[(df_meta_f['study_id'] == int(k)) &
(df_meta_f['series_id'] == int(s))]['series_description'].iloc[0])
except:
print("Failed on", s, k)e. Ví dụ kết quả
meta_obj[list(meta_obj.keys())[1]]{'folder_path': '/kaggle/input/rsna-2024-lumbar-spine-degenerative-classification/train_images/1972129014',
'SeriesInstanceUIDs': ['2898623075', '3324327485', '3203550406'],
'SeriesDescriptions': ['Sagittal T2/STIR', 'Axial T2', 'Sagittal T1']}Nghĩa là:
- Bệnh nhân có ID
1972129014có 3 series, lần lượt là:- 2898623075: Sagittal T2/STIR
- 3324327485: Axial T2
- 3203550406: Sagittal T1
4. Hiển thị ảnh MRI của một bệnh nhân
Truy xuất meta thông tin của một bệnh nhân
patient = train.iloc[1]
ptobj = meta_obj[str(patient['study_id'])]
print(ptobj){
'folder_path': '/kaggle/input/rsna-2024-lumbar-spine-degenerative-classification/train_images/4646740',
'SeriesInstanceUIDs': ['3666319702', '3486248476', '3201256954'],
'SeriesDescriptions': ['Sagittal T2/STIR', 'Sagittal T1', 'Axial T2']
}- folder_path: Thư mục chứa ảnh MRI của bệnh nhân.
- SeriesInstanceUIDs: Danh sách các series (bộ ảnh) cho các kiểu chụp/mặt phẳng khác nhau.
- SeriesDescriptions: Mô tả từng series (ví dụ: Sagittal T2/STIR, Sagittal T1, Axial T2). Đưa dữ liệu ảnh vào cấu trúc dễ thao tác
im_list_dcm = {
'{SeriesInstanceUID}': {
'images': [
{'SOPInstanceUID': ...,
'dicom': PyDicom object
},
...
],
'description': # SeriesDescription
},
...
}Cách thực hiện:
- Với mỗi series của bệnh nhân, lặp qua tất cả các file
.dcm(ảnh MRI lát cắt), lưu lại:- SOPInstanceUID: Mã số ảnh
- dicom: Đối tượng PyDicom chứa toàn bộ dữ liệu ảnh và metadata
im_list_dcm = {}
for idx, i in enumerate(ptobj['SeriesInstanceUIDs']):
im_list_dcm[i] = {'images': [], 'description': ptobj['SeriesDescriptions'][idx]}
images = glob.glob(f"{ptobj['folder_path']}/{ptobj['SeriesInstanceUIDs'][idx]}/*.dcm")
for j in sorted(images, key=lambda x: int(x.split('/')[-1].replace('.dcm', ''))):
im_list_dcm[i]['images'].append({
'SOPInstanceUID': j.split('/')[-1].replace('.dcm', ''),
'dicom': pydicom.dcmread(j) })Hiển thị ảnh của từng series
def display_images(images, title, max_images_per_row=4):
num_images = len(images)
num_rows = (num_images + max_images_per_row - 1) // max_images_per_row
fig, axes = plt.subplots(num_rows, max_images_per_row, figsize=(5, 1.5 * num_rows))
if num_rows > 1:
axes = axes.flatten()
else:
axes = [axes]
for idx, image in enumerate(images):
ax = axes[idx]
ax.imshow(image, cmap='gray')
ax.axis('off')
for idx in range(num_images, len(axes)):
axes[idx].axis('off')
fig.suptitle(title, fontsize=16)
plt.tight_layout()
for i in im_list_dcm:
display_images([x['dicom'].pixel_array for x in im_list_dcm[i]['images']],
im_list_dcm[i]['description'])- Chức năng: Hiển thị các ảnh MRI (dạng lát cắt) cho từng series theo mô tả (ví dụ: “Sagittal T2/STIR”).
- Mỗi lần lặp sẽ hiện toàn bộ ảnh lát cắt của một series (tối đa 4 ảnh trên mỗi hàng, tự động xuống hàng).

Hiển thị tọa độ vùng tổn thương trên ảnh MRI
Dữ liệu về tọa độ tổn thương
- train_label_coordinates.csv chứa các thông tin:
study_id: Mã bệnh nhânseries_id: Mã series (bộ ảnh)instance_number: Số thứ tự lát cắt MRIcondition: Loại tổn thương (ví dụ: Spinal Canal Stenosis)level: Vị trí đốt sống (L1/L2, L2/L3,…)x, y: Tọa độ vùng tổn thương trên ảnh MRI
df_coor = pd.read_csv('/kaggle/input/rsna-2024-lumbar-spine-degenerative-classification/train_label_coordinates.csv')
df_coor.head()| study_id | study_id | series_id | instance_number | condition | level | x | y |
|---|---|---|---|---|---|---|---|
| 0 | 4003253 | 702807833 | 8 | Spinal Canal Stenosis | L1/L2 | 322.831858 | 227.964602 |
| 1 | 4003253 | 702807833 | 8 | Spinal Canal Stenosis | L2/L3 | 320.571429 | 295.714286 |
| 2 | 4003253 | 702807833 | 8 | Spinal Canal Stenosis | L3/L4 | 323.030303 | 371.818182 |
| 3 | 4003253 | 702807833 | 8 | Spinal Canal Stenosis | L4/L5 | 335.292035 | 427.327434 |
| 4 | 4003253 | 702807833 | 8 | Spinal Canal Stenosis | L5/S1 | 353.415929 | 483.964602 |
Hàm hiển thị vị trí tổn thương trên ảnh
def display_coor_on_img(c, i, title):
center_coordinates = (int(c['x']), int(c['y']))
radius = 10
color = (255, 0, 0) # Màu đỏ BGR
thickness = 2
IMG = i['dicom'].pixel_array
IMG_normalized = cv2.normalize(IMG, None, alpha=0, beta=255, norm_type=cv2.NORM_MINMAX, dtype=cv2.CV_8U)
IMG_with_circle = cv2.circle(IMG_normalized.copy(), center_coordinates, radius, color, thickness)
IMG_with_circle = cv2.cvtColor(IMG_with_circle, cv2.COLOR_BGR2RGB)
plt.imshow(IMG_with_circle)
plt.axis('off')
plt.title(title)
plt.show()- Vẽ một vòng tròn đỏ lên vị trí tổn thương trên ảnh MRI (theo tọa độ x, y đã gán nhãn).
- Hiển thị lên màn hình với tiêu đề tùy ý.
coor_entries = df_coor[df_coor['study_id'] == int(patient['study_id'])]
print("Chỉ hiển thị các trường hợp nặng (Severe) cho bệnh nhân này")
for idc, c in coor_entries.iterrows():
for i in im_list_dcm[str(c['series_id'])]['images']:
if int(i['SOPInstanceUID']) == int(c['instance_number']):
try:
patient_severity = patient[
f"{c['condition'].lower().replace(' ', '_')}_{c['level'].lower().replace('/', '_')}"
]
except Exception as e:
patient_severity = "unknown severity"
title = f"{i['SOPInstanceUID']} \n{c['level']}, {c['condition']}: {patient_severity} \n{c['x']}, {c['y']}"
if patient_severity == 'Severe':
display_coor_on_img(c, i, title)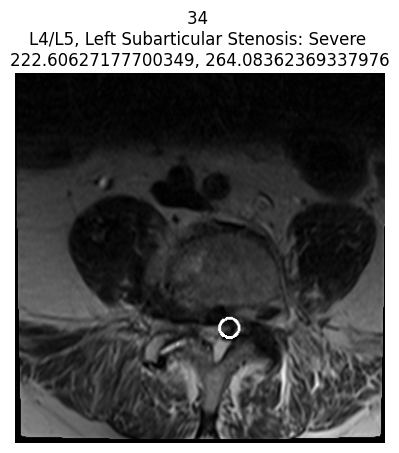
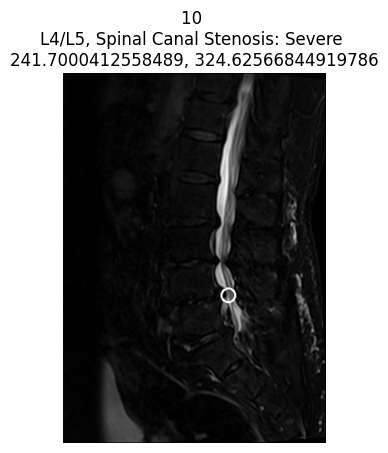
- Lọc ra tất cả các vùng tổn thương được gán nhãn cho bệnh nhân đang xét.
- Với mỗi vùng tổn thương, tìm ảnh MRI tương ứng (dựa trên
series_idvàinstance_number). - Kiểm tra mức độ tổn thương (severe hay không).
- Nếu là Severe, vẽ vòng tròn lên vị trí tổn thương và hiển thị ảnh.